Clone virulento de Pseudomonas aeruginosa KPC-2-positivo na Patagônia Chilena
Análise genômica e fenotípica de Pseudomonas aeruginosa KPC-2/ST654 isolado na Patagônia Chilena revela elevado potencial virulento e resistência a múltiplos antibióticos, destacando a importância da vigilância epidemiológica.
7/30/20254 min read
Estudo detalha resistência e virulência do clone ST654-O4 resistente a carbapenêmicos, evidenciando sua disseminação e impacto na região
Pseudomonas aeruginosa é um patógeno humano oportunista, responsável por uma série de infecções graves, agudas e crônicas, principalmente em ambientes hospitalares. Com alta taxa de mortalidade, este microrganismo tem sua importância reforçada pela crescente resistência a antibióticos, especialmente aos carbapenêmicos, considerados agentes críticos para o tratamento de infecções bacterianas multirresistentes.
Dentre as linhagens de P. aeruginosa, destacam-se os chamados clones de alto risco, classificados com base em sua dominância epidemiológica, virulência e resistência a múltiplos medicamentos. No cenário mundial, o clone ST654-O4 figura entre os “top 10” clones de alto risco, especialmente na América do Sul, onde tem sido identificado em surtos e infecções hospitalares graves.
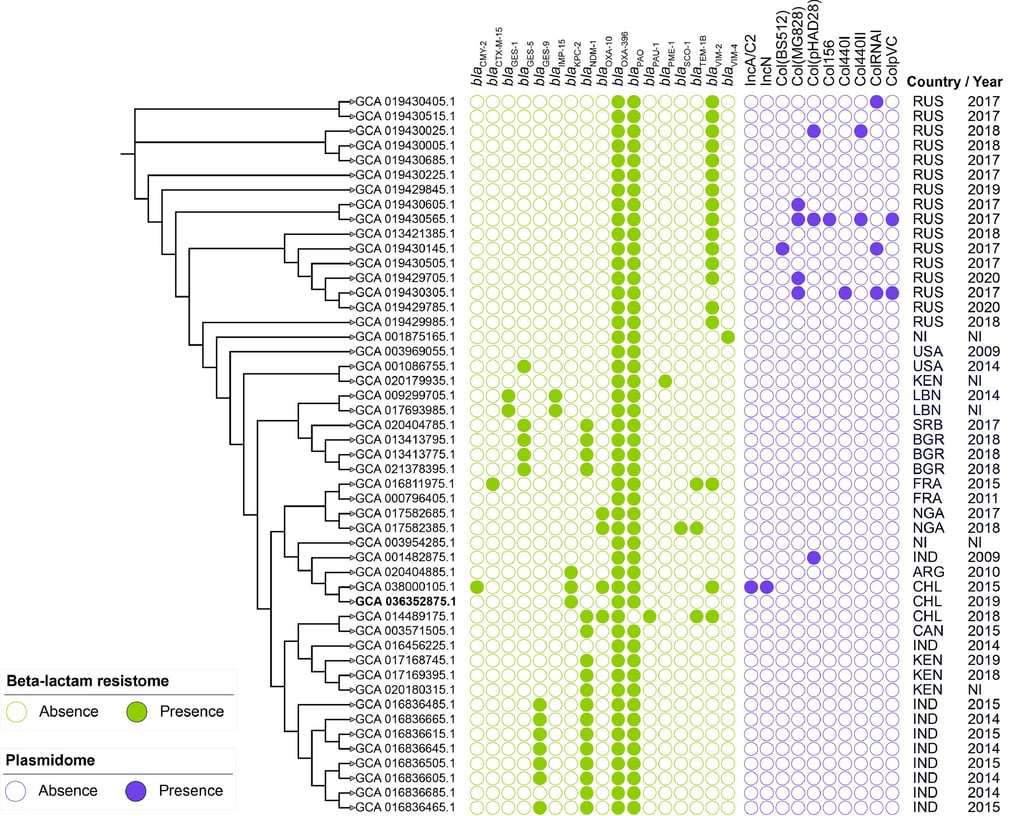
Figura 1: Árvore filogenética enraizada no ponto médio, construída com base no polimorfismo de nucleotídeo único (SNP) de 49 cepas de Pseudomonas aeruginosa ST654 isoladas globalmente de humanos. A árvore é acompanhada pela presença de genes codificadores de beta-lactamases, representados por quadrados verde-claro, replicons plasmidiais indicados por círculos azuis, e informações epidemiológicas, como país e ano de isolamento, quando disponíveis. A sigla NI indica dados não informados. Os países foram identificados conforme o código ISO 3166-1 Alpha-3, com as seguintes abreviações: ARG (Argentina), BGR (Bulgária), CAN (Canadá), CHL (Chile), FRA (França), IND (Índia), KEN (Quênia), LBN (Líbano), NGA (Nigéria), RUS (Rússia), SRB (Sérvia) e USA (Estados Unidos da América). A topologia da árvore e a barra de escala foram geradas automaticamente pelo software iTol v6, utilizando parâmetros padrão, sendo que a escala representa o número de substituições por sítio ao longo dos ramos.
Um estudo recente publicado na Revista de Resistência Antimicrobiana Global investigou a caracterização fenotípica e genômica de uma cepa de P. aeruginosa KPC-2/ST654 isolada na região da Patagônia Chilena. Esta cepa foi obtida de um paciente do sexo masculino, 76 anos, com histórico clínico complexo, incluindo hipertensão, insuficiência renal, uropatia obstrutiva, uso de anticoagulantes e portador de cateter urinário, que foi internado em unidade de terapia intensiva após desenvolver infecção do trato urinário associada a choque séptico e pielonefrite aguda.
A bactéria foi isolada a partir de cultura de urina e identificada por sistema VITEK® 2 GN, apresentando perfil multirresistente, especialmente à carbapenêmicos. A análise genômica de sequenciamento completo revelou a presença do gene bla_KPC-2, responsável pela produção da carbapenemase KPC-2, conferindo alta resistência a este importante grupo de antibióticos. Além disso, a cepa carregava múltiplos genes de resistência, como OXA-396, PAO, aph(3')-Ib, entre outros.
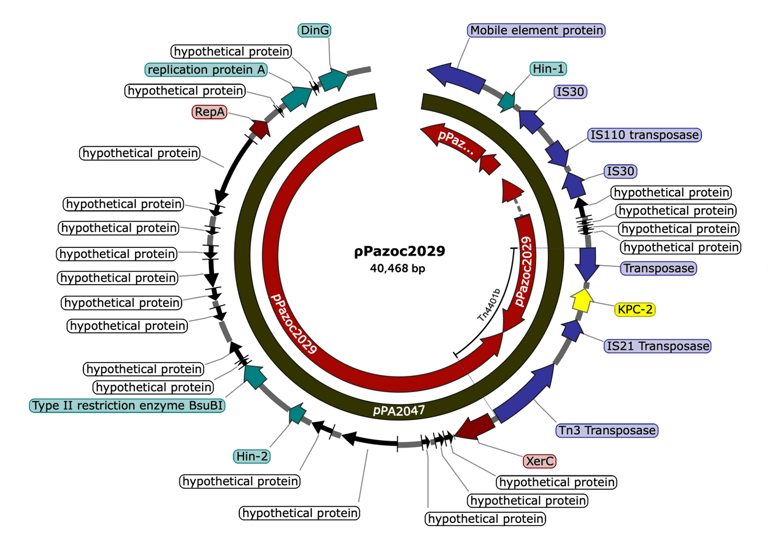
Reconstrução in silico do plasmídeo portador do gene blaKPC-2 em Pseudomonas aeruginosa Pazoc2029, utilizando como referência o plasmídeo pPA2047 (número de acesso GenBank: MN082782, novembro de 2019). A montagem final do plasmídeo reconstruído apresentou 100% de identidade e 93% de cobertura em relação ao pPA2047. O contig contendo o gene blaKPC na cepa Pazoc2029 possui 40.668 pares de bases e alinhou-se com o plasmídeo pPA2047, que possui 43.660 pares de bases.
No que diz respeito à virulência, foram identificados genes associados aos fatores exoenzimáticos exoS, exoY e exoT, conhecidos por contribuírem para a patogenicidade do microrganismo. Para avaliação funcional, o modelo de infecção com larvas de Galleria mellonella foi utilizado. Os resultados indicaram mortalidade de 100% das larvas em apenas 19 horas após a infecção com a cepa ST654, confirmando seu alto potencial virulento. Para comparação, outras cepas de P. aeruginosa de diferentes clones mostraram tempos variados para atingir mortalidade completa, reforçando a agressividade do clone ST654.
Análises filogenômicas mostraram que o clone ST654 KPC-2 tem circulação ativa entre o Chile e a Argentina desde pelo menos 2010, sugerindo uma disseminação regional consolidada. A estreita relação genética entre isolados destas regiões, evidenciada por diferenças mínimas no polimorfismo de nucleotídeo único (SNP), reforça a hipótese de circulação contínua e possível transferência entre ambientes hospitalares dos dois países.
Historicamente, a primeira identificação de P. aeruginosa produtora de KPC-2/ST654 no Chile ocorreu em 2015 na cidade de Santiago, em um paciente internado em UTI. Desde então, relatos indicam que este clone vem se expandindo, principalmente em áreas do sul do Chile, incluindo a Patagônia, com alto impacto clínico devido à resistência a antibióticos de última geração e elevada virulência.
Este cenário apresenta uma preocupação significativa para a saúde pública, pois o controle de infecções causadas por bactérias multirresistentes requer medidas de vigilância epidemiológica rigorosas, estratégias efetivas de prevenção e o desenvolvimento contínuo de alternativas terapêuticas. O estudo reforça a necessidade de monitoramento ativo do clone ST654 e outras linhagens emergentes, para evitar surtos e garantir a eficácia dos protocolos de tratamento.
Em conclusão, o clone Pseudomonas aeruginosa KPC-2/ST654-O4 identificado na Patagônia Chilena demonstra elevada resistência a carbapenêmicos e potencial virulento significativo, configurando-se como uma ameaça crescente na região. A vigilância genômica e epidemiológica contínua é essencial para detectar, controlar e mitigar a disseminação desta linhagem de alto risco.
Referência do artigo: VÁSQUEZ-PONCE, Felipe et al. Potencial virulento de Pseudomonas aeruginosa exoS+ KPC-2-positivo pertencente ao clone de alto risco ST654-O4 identificado na Patagônia Chilena. Revista de Resistência Antimicrobiana Global, 2025. DOI: https://doi.org/10.1016/j.jgar.2025.07.023.


Lucas W.S de Sousa, Biomédico Microbiologista editor e idealizador da página Microbiologia Clínica Brasil.
